こんばんは,@PKです.
前回の記事で,HCSの画像データをCellProfilerで解析⇒Z-Scoreを可視化するという取り組み①を紹介しました.
今回は,以下ワークフロー②の部分(interactive Heatmapを使った可視化と選んだデータの画像を表示)を紹介していきます.
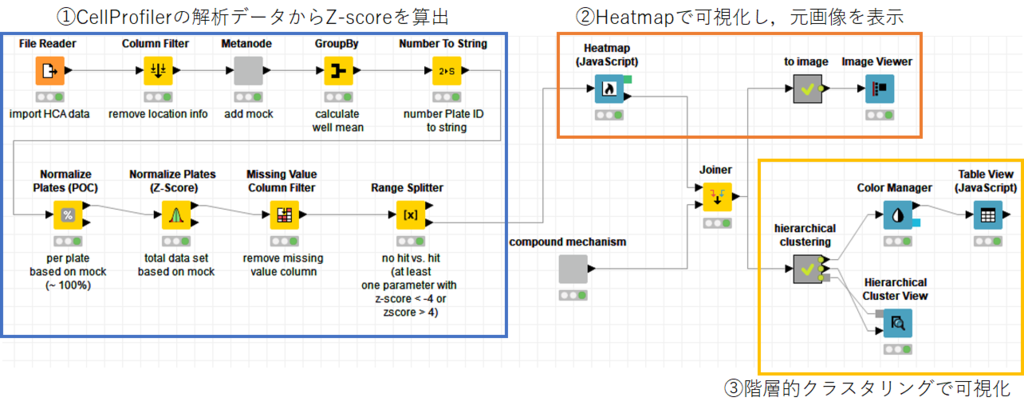
Heatmapについて
まずは,Heatmapの表示についてです.
①の最後に,「RangeSplitter」ノードを使い,特徴量のZ-Scoreがmock(ネガティブコントロール群)と比べて4以上変動している化合物(215/384化合物)だけを抜き出してきました.
ここで紹介するHeatmapは,KNIME JavaScript Views の一つでインタラクティブな操作をすることができます.
Heatmap (JavaScript) — NodePit
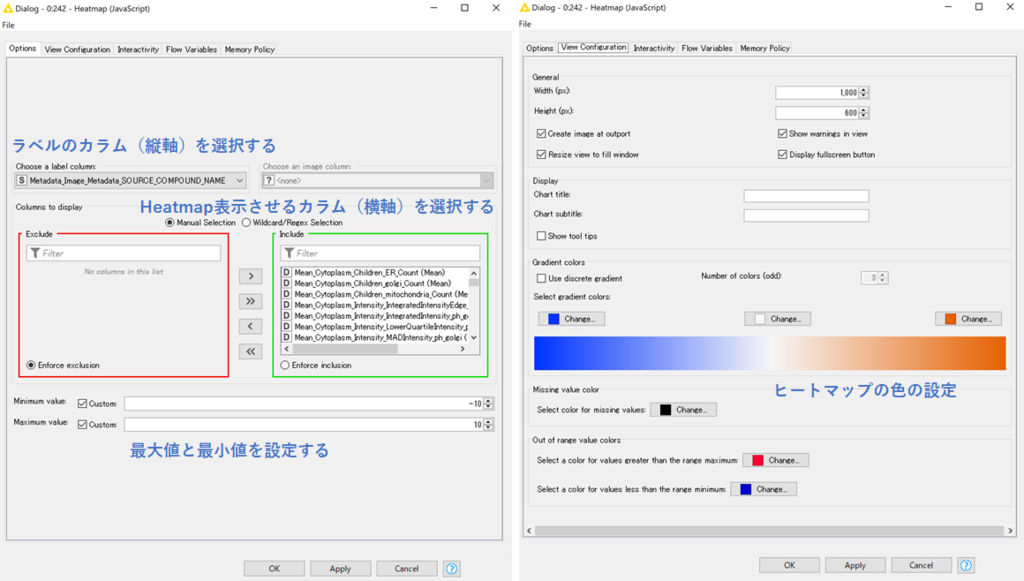
使い方は他のビジュアライゼーションノードと同じで,設定画面からラベルのカラム(縦軸)やHeatmap表示させるカラムを選択します.
今回は,各化合物ごとに,CellProfilerの各特徴量(Z-Score)を表示しています.
また,Heatmapに表示させる値の最大値は±10と設定し,それ以上の値は最大値と同じ色で表示しています(今回は便宜的にこのようにしました…).
「interactive views : heatmap」でheatmapを表示させると以下のようになります.
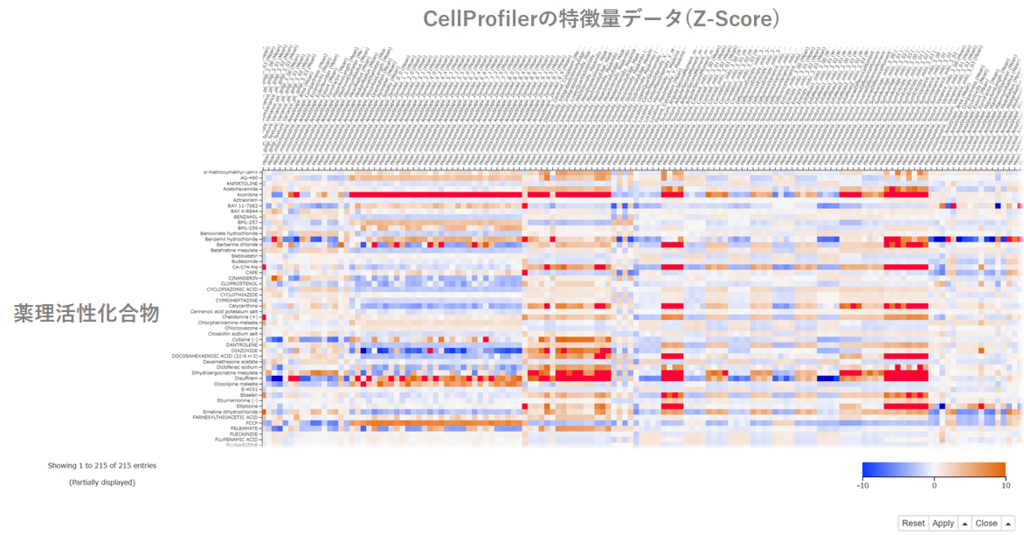
interactive heatmapなので,以下の動画のようにぐりぐりと図を動かしたり,データを選択することことができます.
興味がある化合物があった場合は,「Apply」をクリックすることで選択した情報が次のノードへ受け渡されます.
heatmap上で選択して「Apply」した化合物については,「B(Boolean) Selected (heatmap)」という新しいカラムに結果が出力されます.

画像データの取得
続いて,選択した化合物について細胞の画像データを取得するフローを紹介します.
このフローは,以下のように化合物名に紐づけられている画像ファイル名からローカルの画像データを「Image reader」で読み込み,「Image Viewer」で表示する,という簡単なものです.
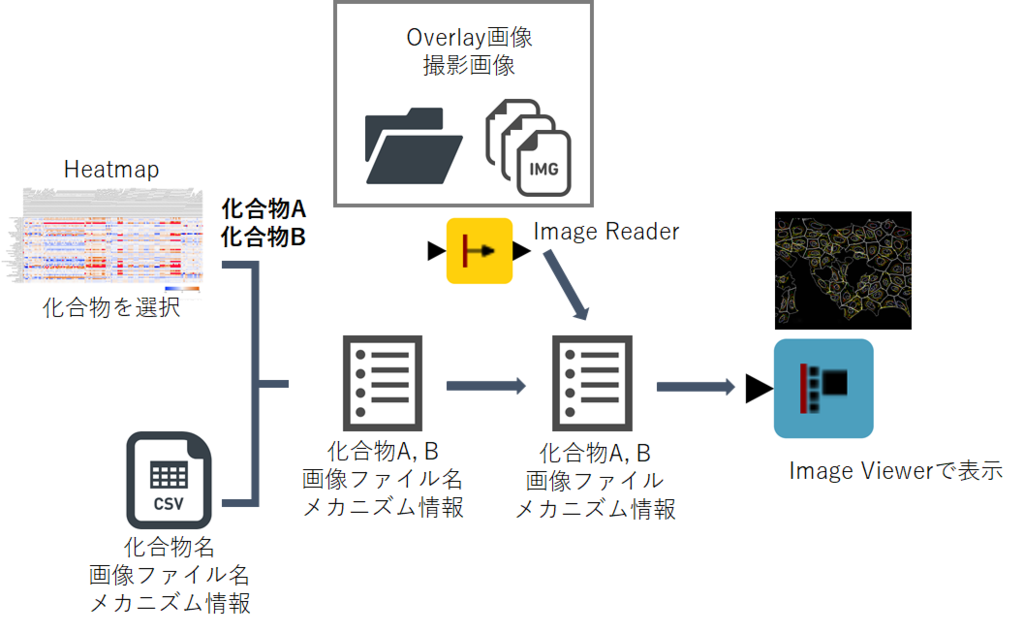
とはいえ,前処理の部分が少し長いので,細かい部分はメタノードでまとめてしまっていました.
以下にメタノードの中身のワークフローを示しています.
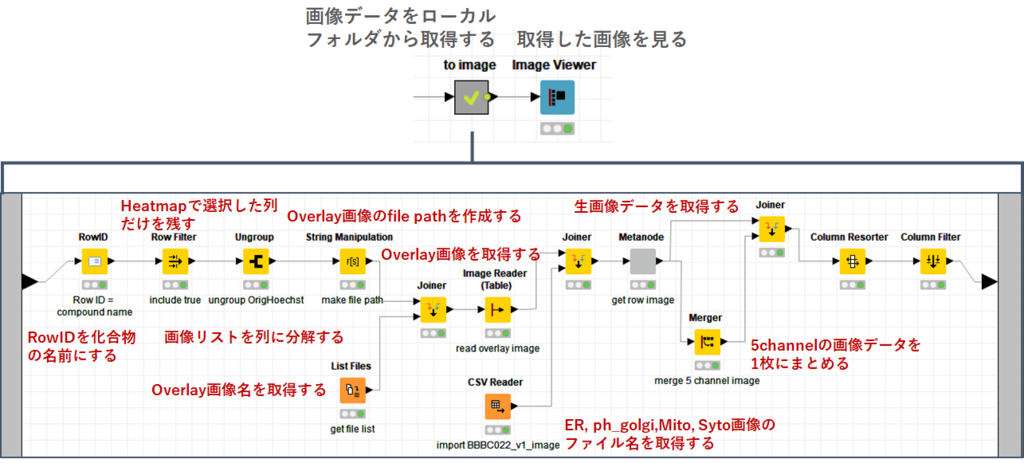
単純なノードの組み合わせなのですが,少し処理が長いのは自分の技術不足なのでご容赦ください…
それぞれのノードでどのような処理をしているかを説明していきます.
ただし,Overlay画像取得と生画像取得はほとんど同じ操作なので,ここではOverlay画像取得に絞って紹介していきます.
まず,「RowID」ノードで化合物名のカラムをIDに設定し直した後に,「RowFilter」でheatmapで選択した化合物だけを抜き出します.
これは「Selected (heatmap)」カラムのTrueの部分だけを選択すればOKです.
続いて,「Ungroup」で画像ファイル名のリスト(ヘキスト染色のファイル名)を列ごとに分解しています.
元々の画像のファイル名のリストは化合物のメカニズム抽出の際に作っておきました(省略してすみません…)
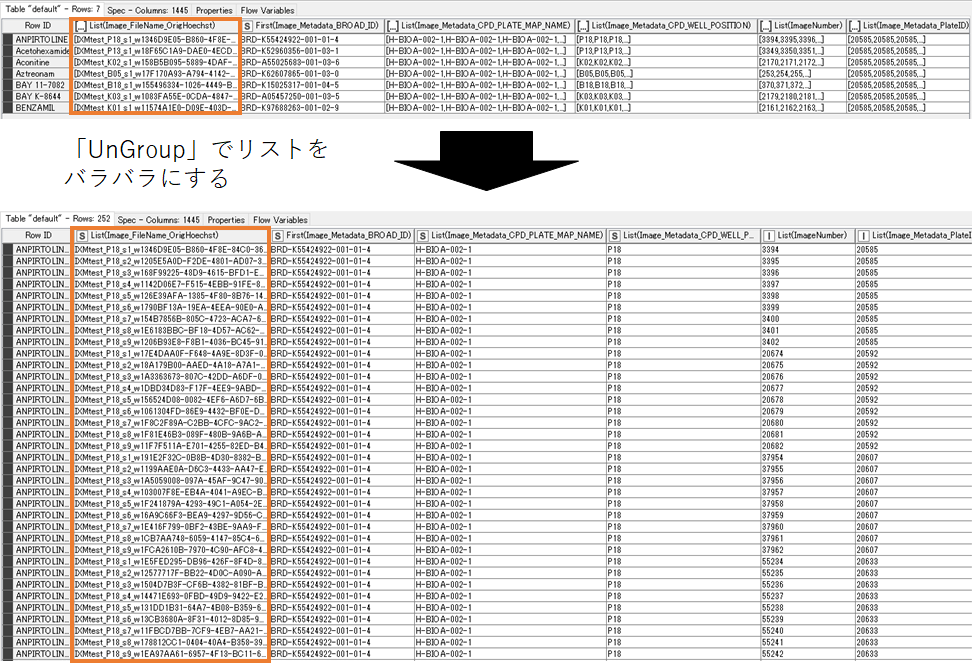
「UnGroup」ノードの使い方は,す さんのブログがわかりやすいです.
【KNIME】ノード紹介:cell splitterとUngroupのコラボ 〜文字列を縦方向に分割したい〜 - 非プログラマーのためのインフォマティクス入門。(仮)
続いて「String Manipulation」ノードで,取得したヘキストの画像ファイル名を基にしてファイルパスを作成します.
今回はOverlay画像を取得したいのですが,これはCellProfiler解析の際にヘキスト染色画像の上に各オブジェクトの重ね合わせをしています.
そのため,Exportされたファイル名は「各視野のヘキスト画像のファイル名+.png」となっています.
【参考】CellProfilerの「SaveImages」モジュール↓
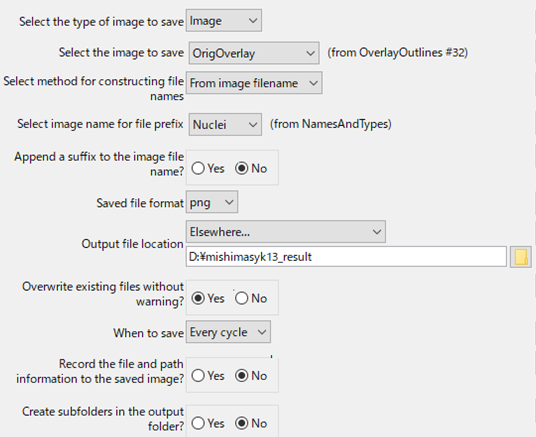
保存されたファイルがある場所を指定して,ファイルパスを作成しました.

続いて「ImageReader」ノードで指定された画像ファイルを読み込んでいきます.
何故か「String Manipulation」ノード直接ではダメだったので(理由はわからないです…)一度「List Files」ノードと「Joiner」でつないだ後に「ImgeReader」ノードで画像を読み込みました.
その結果,以下のように各列(視野)ごとに画像ファイルが読み込まれていることがわかります.
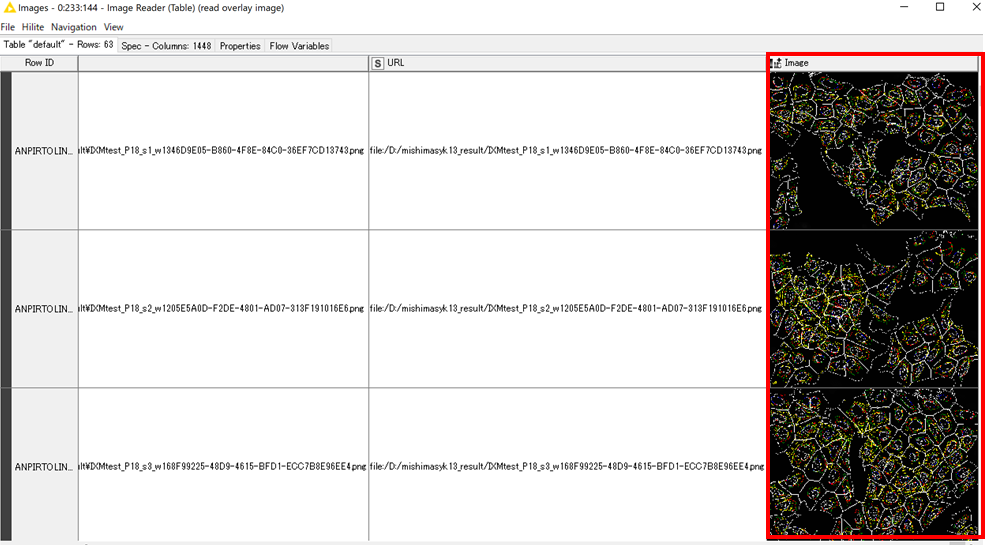
生画像の取得後(今回は割愛)必要なカラムだけを選択したら,最後に「ImageViewer」で画像データを表示させます.
「ImageViewer」では以下のように画像データを拡大・縮小したり,表も同時に見ることができます.
youtu.be
終わりに
今回のワークフローで,CellProfilerの画像解析の結果を可視化したり,そこから画像データを取得することがKNIMEを使うとできることを紹介しました.
CellProfilerとKNIMEはどちらもオープンソースソフトウェアですが,このように画像解析においては非常に協力なツールになると思います.
今後も引き続きこれらの使い方について紹介できるように勉強していきますのでよろしくお願いします.