こんにちは,@PKです.
前回の記事でCellProfilerのExample pipelineを紹介しました.
この記事では,CellProfilerを使って核染色,細胞質染色,PH3(リン酸化ヒストンH3)を染色した画像データから,以下のように「核」「細胞」「PH3」オブジェクトを認識して各領域での蛍光強度やエリア値などを算出しました.
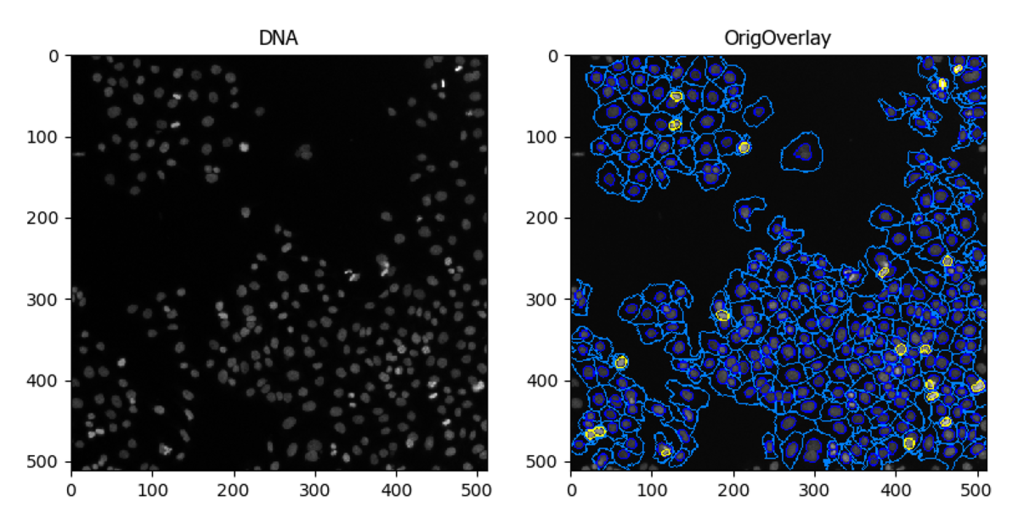
今回は,CellProfilerの解析から得られた結果をKNIMEで可視化してみたので紹介しようと思います.
この記事を書くきっかけは,@Pen先生のブログでChemoinformaticsのデータをKNIMEを使って可視化する記事を読んだことです.
Make interactive plot with Knime #RDKit #Chemoinformatics #Knime | Is life worth living?
この記事を見て是非自分でもやってみたいと思い,HCAのデータを使って同じような可視化を行いました.
まずは,HCAのデータをシンプルに可視化しよう考えました.
画像データだけでは,細胞の位置は定性的にしかわかりません.しかし,CellProfilerの解析(high content analysis)によって細胞の位置情報を定量化することができるので,以下の図のようにKNIME上で画像データの細胞の位置情報を再現するようなScatter plotをかくことができます.

KNIMEのフローは以下の通りです.
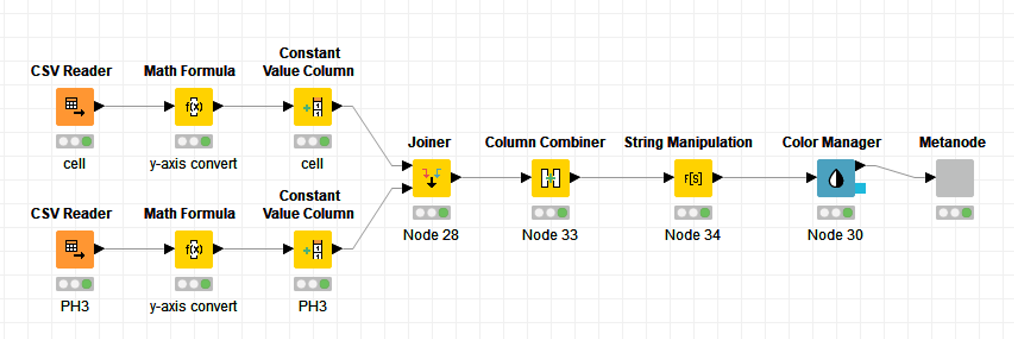
まずは,CellProfilerの解析で得られたCSVファイルを読み込みます.
各列には,抽出した特徴量やオブジェクトの番号が,各行はそれぞれの細胞を示しています.
細胞は視野内に290個あるので,290行のデータがあります.

上図は「Cell」と「PH3」の2つのファイルを読み込んだ結果を示していますが,「Cell」と「Nuclei」,「PH3」と「Nuclei」はそれぞれ紐づいていたので,「Parent Nuclei」を使って2つのデータを紐づけることができそうです.
データの可視化までは単純作業なので簡単に説明します.
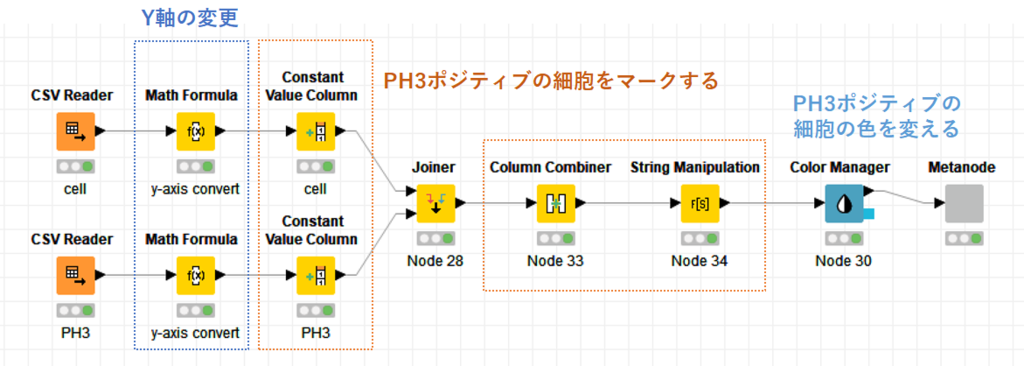
Y軸の変更
CellProfilerで取得した「Cell」の特徴量の中には,各細胞のWell内のX座標,Y座標といった位置情報もあります.しかし,CellProfilerではY軸は上がゼロだったのですが,KNIMEでscatter plotを書くときは原点は左下なので,グラフにすると上下逆になってしまいます. これを「Math Formula」を使って修正しました.
PH3ポジティブの細胞をマーク
「Constant Value column」で「Cell」と「PH3」のカラムをそれぞれのデータに追加しました.
その後「Joiner」で2つのデータを合わせ,「Column combiner」と「String manipulation」を使ってPH3ポジの細胞は「Cell-PH3」,PH3ネガの細胞は「Cell」と名前を付けました.
PH3ポジティブの細胞の色を変える
「Color manager」というノードを使うと,各行ごとに色を付けることができます.ここでは,「Cell」と「Cell-PH3」を色でわけることにしました.
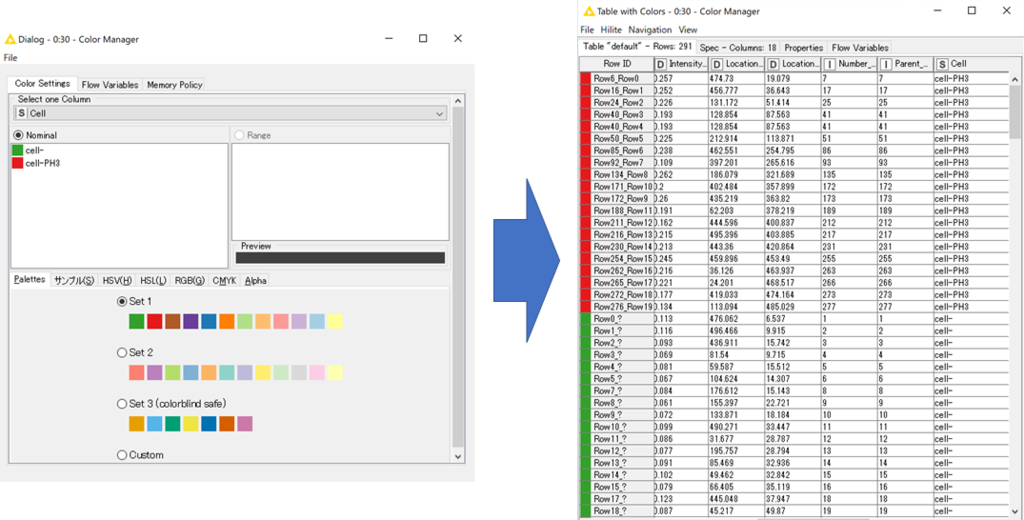
最後にデータの可視化のために作成したMetanodeについて説明します.
「右クリック」⇒「Wrapped Metanode」⇒「Open」でMetanodeを開くと,右下図のようなノードが格納されていることがわかります.
色々なデータの解析や可視化ができますが,今回は「Scatter Plot」,「Table view」,「Conditional Box Plot」を使いました.
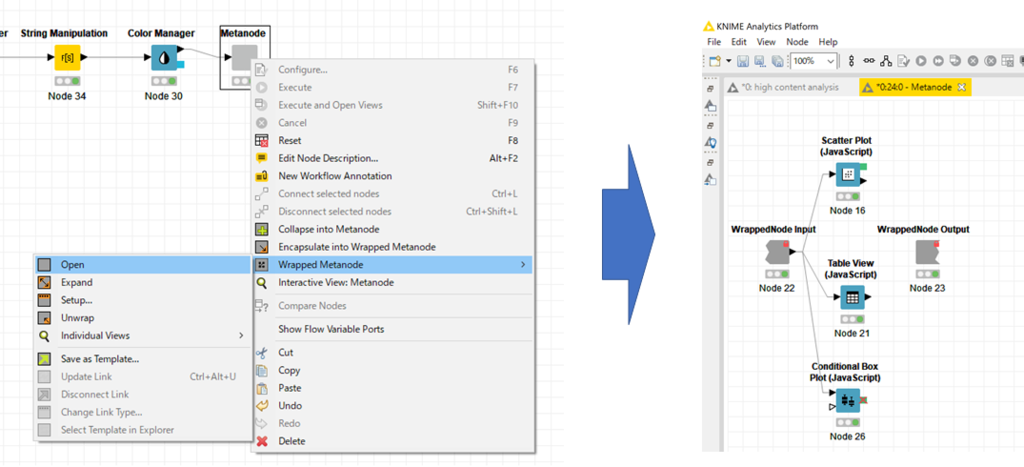
ちなみに,Metanodeを作成するときは,格納したいノードを選択して,「右クリック」⇒「Encapsulate into Wrapped Metanode」で作ることができます.
また,以下のように「Node」⇒「Wrapped Metanode Node Usage And Layout」から,データの表示の仕方も変更することができます!
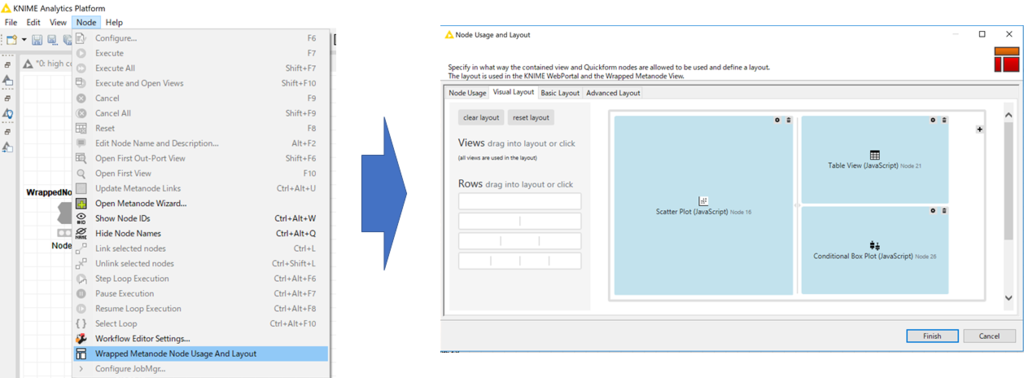
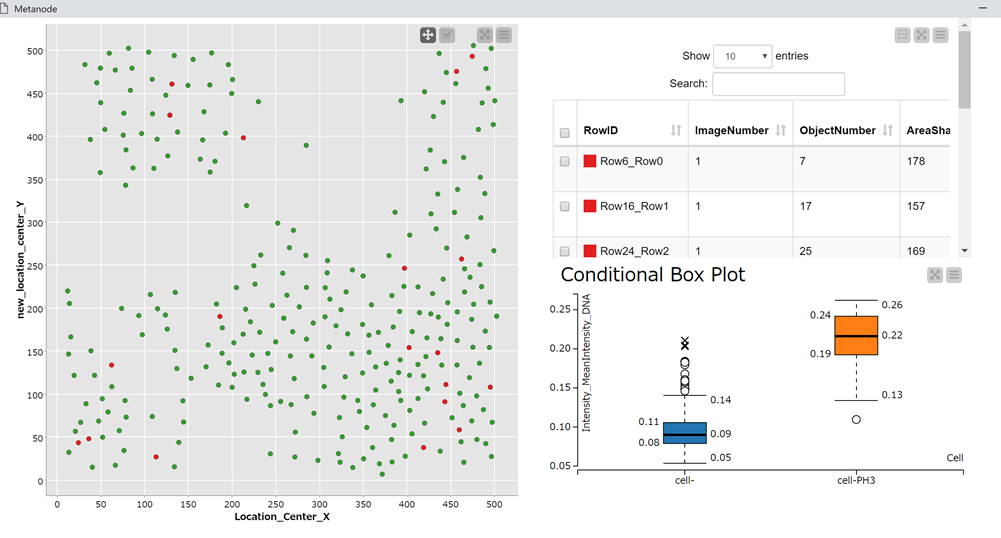
レイアウトも整えたので,実際にMetanode のinteractive viewsを開くと,以下のように,レイアウト通りのグラフや表を出力することができました.
「Scatter Plot」ではX軸とY軸に,各細胞のWell内のX座標,Y座標を使用し,PH3ポジとネガの細胞ごとにで色分けをしました.
また,「Conditional Box Plot」ではPH3ポジとネガの細胞ごとのDNAの平均蛍光強度を表示しています.
Scatter plotやBox plotも簡単に描くことができるので,KNIME大変便利です.
せっかくなので,interactive viewsでどのようにデータを見ることができるか動画で示したいと思います. (pen先生の真似をしました…!)
これらのノードを使うと,単純にグラフを書くだけでなく,可視化されたデータを触って解析することができます.
動画で示していますが,特定の細胞集団の特徴量がどうなっているかなどを簡単に見ることができます.
こんな可視化までKNIMEでできるとは知らなかったので,勉強になりました. 実はCellProfilerのKNIMEノードもあるので,KNIME上で「CellProfilerで画像解析」⇒「データ解析(多変量解析とか)」⇒「ビジュアライゼーション」まで一通りできてしまうみたいですので,引き続きKNIME & CellProfilerの使い方を習得していきます.