先日行われたMishima.syk #12の勉強会で,KNIMEを使って外部DBからデータを取得するということをお話しさせていただく機会をいただきました.
皆さんからいろんなアドバイスをいただくことができて非常に良い機会でしたので,引き続き学び続けられたら,と思っています.
今回は,上記勉強会でも発表した,薬理活性化合物の構造データからChEMBLデータベースを通じてターゲット分子を予想するということをご紹介いたします.
化合物の構造や名前に関する情報から,その化合物がターゲットとする分子の情報を取得したい場合,化合物が一つでしたら手作業で検索をしてターゲットと想定されている分子情報を取得できると思います.
しかし,複数の化合物に対してターゲット分子を調べる場合は調査する手間と時間がかかります.
そこで,ChEMBLというデータベースを使えば,分子の構造情報などからターゲット分子の情報を取得できます.
ChEMBLデータベースとは?
- https://www.ebi.ac.uk/chembl/
- 医薬品及び開発化合物のデータベース
- 無料で検索&ダウンロード可能
- ターゲットやアッセイ情報も紐付けされている
- ChEMBL Web ServicesのページにAPIを利用したデータ取得の詳細が書かれています.
こちらの使い方は下記動画を参考にしてください
- ChEMBLを使って医薬品候補となる化合物について調べる
ChEMBLで検索をする際に,構造データと一対一で対応しているInChIkeyを取得し,情報を取得します.
KNIMEのフローは以下のようになります.
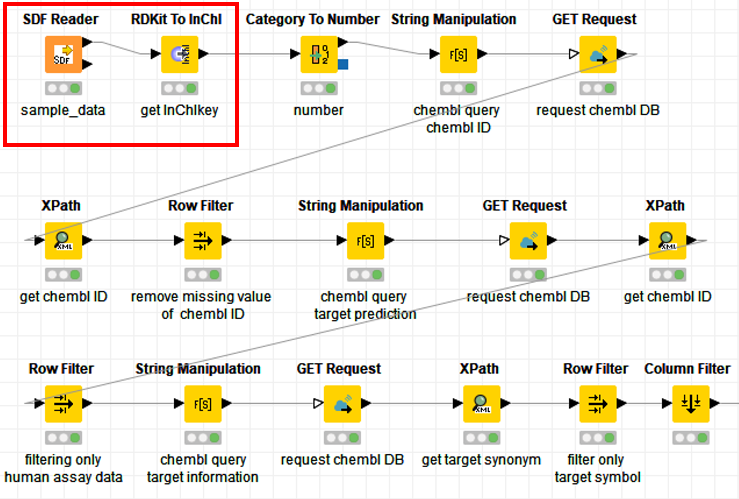
まず,化合物の構造データ(SDF file)をSDF readerで読み込み,続いてRDKit To InChI というノードで構造情報をInChI keyに変換します.
上記ノードがない場合は 「File」⇒ 「Install KNIME Extensions…」から検索をしてノードをインストールすることができます.
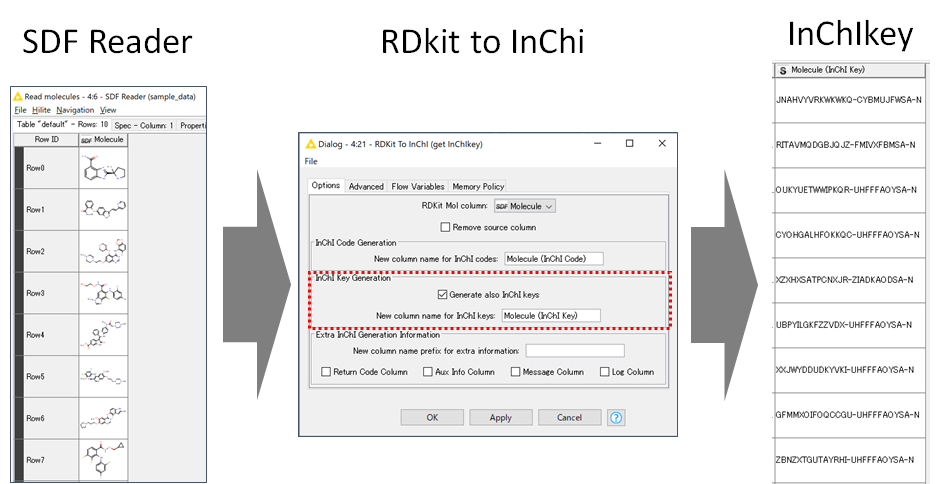
「SDF reader」でSDF fileを読み込むと,以下の図のように化合物の構造情報を読み込めていることがわかります. ここから,「RDkit to InChI」ノードを使ってInChI Codeを生成します. この時,「generate also InChI Keys」にチェックを入れるとInChI Keysを取得することができます. 下記より対応したInChI Keyを取得できていることがわかります.
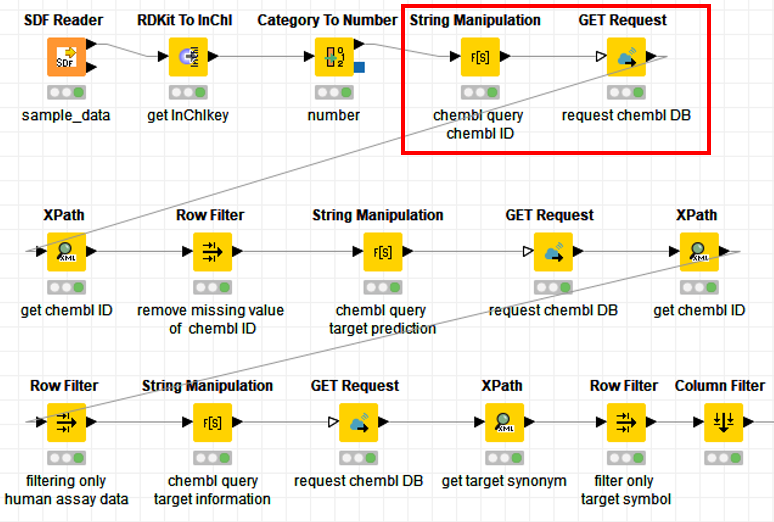
続いて,得られたInChI KeyからChEMBLデータベースにアクセスをして,Chembl IDを取得します.
データベースから情報を取得する流れは,「XML形式」でのデータ取得の場合,
基本的にはString manipulation ⇒ GET Request ⇒ Xpathの繰り返し
と考えてよいと思います.
また,今回はXML形式で取得しましたがJSON形式の場合はXPathではなく,JSON Pathで取得できます.
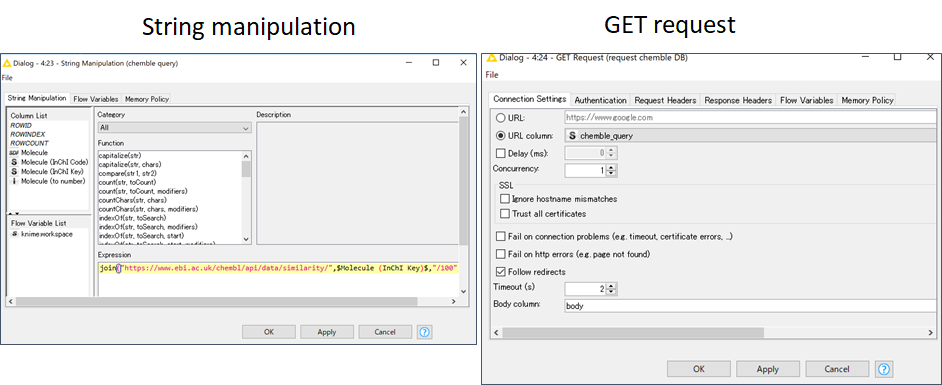
String manipulationで Chembl IDを取得するURLを作成します.
join("https://www.ebi.ac.uk/chembl/api/data/similarity/",\$Molecule (InChI Key)$,"/100")
GET requestでは生成したURL columnを選択するだけです.
リクエストが多い場合は,Delayで連続するリクエスト間の時間を調整することができます.
InChI Keyは完全に一致している化合物データを取得したかったので,similarity = 100で検索をかけています.
続いて,XPathの部分についてです.
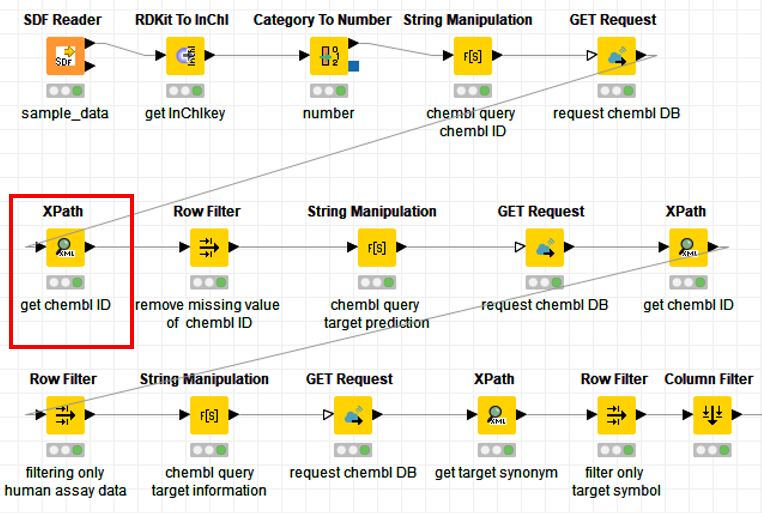
XMLのデータから,必要なデータだけを選んで抽出します.
今回はChembl IDが欲しいのでその部分を抜き出します.
Xpath query : /response/molecules/molecule/molecule_hierarchy/molecule_chembl_id
それぞれの化合物に一対一で対応しているので,「Type」の部分は「String(Single Cell)」にします.
この操作でChEMBL IDを取得することができました.
この後,以下の図のように,
String manipulation ⇒ GET Request ⇒ Xpathを繰り返して化合物と相互作用すると想定されているターゲット分子と,そのターゲット分子名を取得していきます.
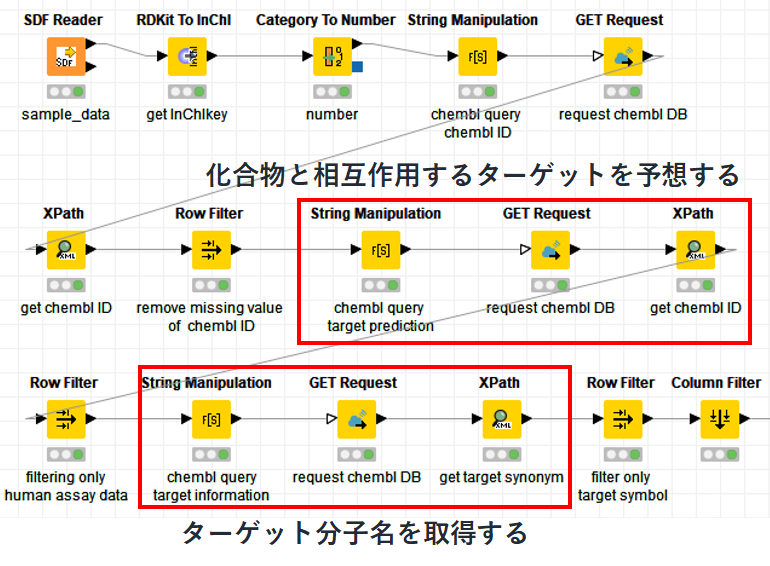
同様の操作なので詳細は割愛しますが,それぞれのString Manipulationで以下のようにデータを取得するURLを作成します.
String manipulation (target prediction)
join("https://www.ebi.ac.uk/chembl/api/data/target_prediction?molecule_chembl_id=",\$molecule_chembl_id$)
Stiring manipulation (target information)
join("https://www.ebi.ac.uk/chembl/api/data/target/",\$target_chembl_id$)
最後に取得したデータの一部をお示しします.
化合物名,ターゲットと言われている分子,今回ChEMBLの「Target Prediction」で予想された分子を以下の表でそれぞれ示しています.

一般的に主のターゲットと報告されている分子以外にも,非常に多くの分子が「Target Prediction」の結果得られました.
もちろん,化合物,特にキナーゼ阻害剤などは特異的阻害剤と言われているものでも非常に様々なターゲットに触っている場合が多いため,このような結果となっている可能性もあります.
上記化合物の詳細なプロファイルまでは確認していないので,今後得られた結果の妥当性を考えていければと考えています.